¿Cómo se contraen los músculos? ¿Qué moléculas son necesarias para que un tejido cambie su forma?
El músculo es un tejido contráctil especializado que les da una característica distintiva de los animales. Los cambios en la longitud del músculo apoyan una exquisita variedad de movimientos de animales, de la destreza de los tentáculos del pulpo y las ondas peristálticas de Aplysia a la coordinación precisa de linebackers y ballerinas. ¿Qué mecanismos moleculares dan lugar a la contracción muscular? El proceso de contracción tiene varios pasos clave, que se han conservado durante la evolución a través de la mayoría de los animales.

Figura 1: Un músculo gastrocnemio (gemelo), con patrón de rayas de sarcómeros
La vista de un músculo gastrocnemio del ratón (pantorrilla) bajo un microscopio. Los sarcómeros son de color verde artificial, y aparecen como rayas apilados horzontal de longitudes similares. (Barra de escala blanca = 25 micras).
Cuando las células musculares se observan bajo el microscopio, se puede ver que contienen un patrón de rayas (estrías). Este patrón está formado por una serie de unidades base llamado sarcómeros que están dispuestos en un patrón apilado en todo el tejido muscular (Figura 1). Puede haber miles de sarcómeros en una sola célula muscular. Los sarcómeros son altamente estereotipados y se repiten a lo largo de las células musculares, y las proteínas dentro de ellos pueden cambiar de longitud, lo que hace que la longitud total de un músculo pueda cambiar. Un sarcómero individual contiene muchos filamentos paralelos de actina (finas) y miosina (gruesos) (fig. 1A). La interacción de las proteínas de miosina y actina es la base de nuestra comprensión actual del acortamiento del sarcómero. ¿Cómo se produce esta reducción? Tiene algo que ver con una interacción deslizante entre la actina y la miosina.
La teoría del filamento deslizante
En 1954, los científicos publicaron dos documentos innovadores que describen las bases moleculares de la contracción muscular. Estos documentos describen la posición de los filamentos de la miosina y la actina en varias etapas de contracción de las fibras musculares y propusieron cómo esta interacción produce la fuerza contráctil. Usando microscopía de alta resolución, AF Huxley y R. Niedergerke (1954) y HE Huxley y J. Hanson (1954) observaron cambios en los sarcómeros del tejido muscular acortado. Ellos observaron que una zona de la configuración repetida del sarcómero, la «banda A,» se mantuvo relativamente constante en longitud durante la contracción (figura 2A). La banda A contiene filamentos gruesos de miosina, lo cual sugiere que los filamentos de miosina centrales se mantienen constantes en longitud, mientras que otras regiones del sarcómero se acortan. Los investigadores señalaron que la «banda I,» rica en filamentos delgados hechos de actina, cambia su longitud a lo largo del sarcómero. Estas observaciones llevaron a proponer la teoría del filamento deslizante, que establece que el deslizamiento de la actina pasando sobre miosina genera tensión muscular. Debido a la actina está atada a las estructuras situadas en los extremos laterales de cada sarcómero llamado discos Z, cualquier acortamiento de la longitud de los filamentos de actina daría lugar a un acortamiento del sarcómero y por lo tanto el músculo. Esta teoría se ha mantenido impresionantemente intacta.

Figura 2: Comparación de un sarcómero relajado y contraído
(A) La organización básica de una subregión de sarcómero, que muestra la ubicación centralizada de la miosina (banda A). La actina y los discos Z se muestran en rojo. (B) Un diagrama conceptual que representa la conectividad de moléculas dentro de un sarcómero. Una persona de pie entre dos estanterías (bandas Z) tira de ellas mediante cuerdas (actina). Miosina (M) es análoga a la persona y los brazos de tracción. (Bandas Z también se llaman los discos z.)
Una analogía para el acortamiento del sarcómero
Imagínese que usted está de pie entre dos grandes estanterías llenas de libros. Estos grandes estanterías están a varios metros de distancia y se colocan en los carriles de modo que puedan ser movidas fácilmente. Se le da la tarea de llevar los dos estantes para libros juntos, y permiten usar sólo los brazos y dos cuerdas. De pie centrado entre las estanterías, se tira de las dos cuerdas – uno por cada brazo – que están atadas firmemente a cada biblioteca. De manera repetitiva, se tira cada cuerda hacia usted, la recupera, y luego tira de nuevo. Con el tiempo, a medida que avanza a través de la longitud de la cuerda, las estanterías se mueven juntas y se acercan a usted. En este ejemplo, los brazos son similares a las moléculas de miosina, las cuerdas son los filamentos de actina y las estanterías son los discos Z en que se asegura la actina, que constituyen los extremos laterales de un sarcómero. Igual a la forma en que usted se mantendrá centrado entre las estanterías, los filamentos de miosina permanecen centrados durante la contracción muscular normal (Figura 2B).
¿Qué son los puentes que cruzan?
Un refinamiento importante de la teoría del filamento deslizante consistió en la forma particular en que la miosina es capaz de empujar a la actina para acortar el sarcómero. Los científicos han demostrado que el extremo globular de la proteína de miosina más cercana a la actina, llama la región S1, tiene múltiples segmentos articulados, que se pueden doblar y facilitan la contracción (Hynes et al. 1987; Spudich 2001). La flexión de la región de la miosina S1 ayuda a explicar la forma en que se mueve o «pasea» la miosina a lo largo de la actina. La región de «cola» más delgado y por lo general más largo de la miosina (S2) también exhibe flexibilidad, y gira simultáneamente con la contracción de S1 (Figura 3A).
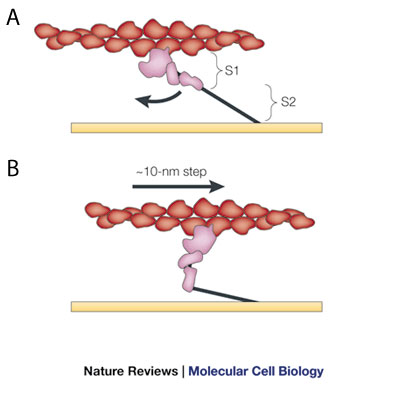
Figura 3: El «golpe de potencia» del modelo de puente de balanceo, a través del movimiento «ciclista» de la miosina-actina
La actina (rojo) interactúa con la miosina, que se muestra en forma globular (rosa) y en forma de filamento (línea de color negro). El modelo que se muestra es el de HE Huxley, modificado para indicar (flecha curvada) la flexión cerca de la mitad del puente transversal de forma alargada (subfragmento 1, o S1) que proporciona el «golpe de potencia». Esta flexión impulsa actina a la derecha a unos 10 nanómetros (nm de paso 10). Las áreas S2 amarran la miosina globular al filamento grueso (línea amarilla horizontal), que permanece en el lugar mientras que los filamentos de actina se mueven. Modificado de Spudich (2001).
Los movimientos de la miosina parecen ser una especie de danza molecular. La miosina avanza hacia adelante, se une a la actina, se contrae, libera la actina, y luego avanza de nuevo hacia delante para unirse actina en un nuevo ciclo. Este proceso se conoce como el ciclismo (bicicleta) de miosina-actina. A medida que el segmento S1 de miosina se une y libera la actina, forma lo que se denominan puentes cruzados, que se extienden desde los filamentos gruesos de miosina a los filamentos finos de actina. La contracción de la región de S1 de la miosina se llama power stroke (golpe de potencia) (Figura 3). La carrera de potencia requiere la hidrólisis de ATP , que se rompe un enlace de fosfato de alta energía y libera energía.
En concreto, esta hidrólisis ATP proporciona la energía a la miosina que pasar por este ciclo: liberar la actina, cambiar su conformación, contraerse, y repetir el proceso de nuevo (Figura 4). La miosina se mantendría vinculado a la actina indefinidamente – haciendo la rigidez del rigor mortis – si las nuevas moléculas de ATP no estuvieran disponibles (Lorand 1953).

Figura 4: Ilustración del ciclo de cambios en la forma de miosina durante los ciclos de puentes cruzados (1, 2, 3 y 4) Hidrólisis de ATP libera la energía necesaria para la miosina para hacer su trabajo. AF: filamentos de actina, miosina MF filamento. Modificado de Goody (2003).
Dos aspectos clave del ciclismo miosina-actina utilizan la energía puesta a disposición por la hidrólisis de ATP. En primer lugar, la acción del alcance de la cabeza S1 de miosina utiliza la energía liberada después de que la molécula de ATP se divide en ADP y fosfato (P). La miosina se une actina en esta conformación extendida. En segundo lugar, la liberación del fosfato faculta a la contracción de la región S1 de miosina (Figura 4).
¿Qué regula el acortamiento del sarcómero?
El calcio y el ATP son cofactores (componentes no proteicos de los enzimas) necesarios para la contracción de las células musculares. El ATP proporciona la energía, tal como se describe más arriba, pero ¿qué hace el calcio? El calcio se requiere para dos proteínas, la troponina y la tropomiosina, que regulan la contracción muscular mediante el bloqueo de la unión de la miosina a la actina filamentosa (Figura 5).
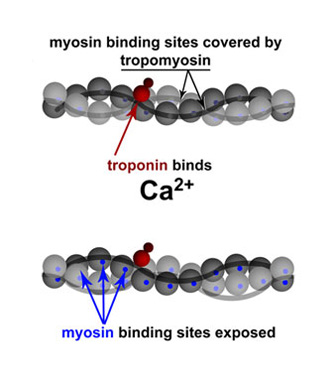
En un sarcómero en reposo, la tropomiosina bloquea la unión de la miosina a la actina. En la analogía superior de los estantes de tracción, la tropomiosina se pondrá en el camino de la mano mientras trataba de sostener la cuerda de actina. Para que la miosina se una a la actina, la tropomiosina debe girar alrededor de los filamentos de actina para exponer los sitios de unión a la miosina. En 1994, William Lehman y sus colegas demostraron cómo la tropomiosina gira estudiando la forma de la actina y la miosina, ya sea en soluciones ricas en calcio o soluciones que contengan calcio bajo (Lehman, Craig, y Vibertt 1994). Mediante la comparación de la acción de la troponina y la tropomiosina bajo estas dos condiciones, encontraron que la presencia de calcio es esencial para el mecanismo de contracción. Específicamente, la troponina (la proteína más pequeña) desplaza la posición de la tropomiosina y la mueve lejos de los sitios de unión a miosina en la actina, para desbloquear de forma efectiva el sitio de unión (Figura 4). Una vez que los sitios de unión a la miosina están expuestos, y si hay presente suficiente ATP, la miosina se une a la actina para comenzar el ciclismo a través del puente. Entonces se acorta el sarcómero y el músculo se contrae. En ausencia de calcio, no se produce esta unión, por lo que la presencia de calcio libre es un importante regulador de la contracción muscular.
Preguntas sin resolver
Entendemos completamente la contracción muscular? Los científicos todavía tienen curiosidad acerca de varias proteínas que influyen claramente en la contracción muscular, y estas proteínas son interesantes porque están bien conservadas a través de las especies animales. Por ejemplo, moléculas como la titina, una proteína «elástica» inusualmente larga y que abarca sarcómeros en los vertebrados, parece unirse a la actina, pero no se conocen bien. Además, los científicos han hecho muchas observaciones de las células musculares que se comportan de maneras que no coinciden con el conocimiento actual de las mismas. Por ejemplo, algunos músculos en moluscos y artrópodos generan la fuerza durante largos períodos, un fenómeno poco entendido a veces llamado «captura-tensión» o histéresis de fuerza (Hoyle, 1969). El estudio de estos y otros ejemplos de cambios musculares (plasticidad) son vías interesantes para la exploración por los biólogos. En última instancia, esta investigación puede ayudarnos a comprender mejor y tratar a los sistemas neuromusculares y comprender mejor la diversidad de este mecanismo en nuestro mundo natural.
Resumen
La contracción muscular ofrece a los animales una gran flexibilidad, lo que permite que se muevan de manera exquisita. Los cambios moleculares que dan lugar a la contracción del músculo se han conservado en la evolución en la mayoría de los animales. Mediante el estudio de los sarcómeros, la unidad básica de control de los cambios en la longitud muscular, los científicos propusieron la teoría del filamento deslizante para explicar los mecanismos moleculares que subyacen a la contracción muscular. Dentro del sarcómero, la miosina se desliza a lo largo de la actina para contraer la fibra muscular en un proceso que requiere ATP. Los científicos también han identificado muchas de las moléculas implicadas en la regulación de las contracciones musculares y comportamientos motrices, incluyendo el calcio, la troponina y la tropomiosina. Esta investigación nos ayudó a aprender cómo los músculos pueden cambiar su manera de producir movimientos.
Referencias y lecturas recomendadas
Clark, M. Milestone 3 (1954): modelo de deslizamiento de filamentos para la contracción muscular. Correderas filamentos musculares.Nature Reviews Molecular Biología Celular 9 , S6-S7 (2008) doi: 10.1038/nrm2581.
Goody, RS El eslabón perdido en el ciclo de los puentes cruzados muscular. Nature Structural Molecular Biology 10 , 773-775 (2003) doi: 10.1038/nsb1003-773.
Hoyle, G. Aspectos comparativos de músculo. Revisión Anual de Fisiología 31 , 43-82 (1969) doi: 10.1146/annurev.ph.31.030169.000355.
Huxley, HE & Hanson, J. Cambios en el cruce de las estrías de los músculos durante la contracción y el estiramiento y su interpretación estructural Nature 173 , 973-976 (1954) doi: 10.1038/173973a0.
Huxley, AF & Niedergerke, R. Los cambios estructurales en el músculo durante la contracción: Interferencia de microscopía de las fibras musculares que viven. Naturaleza 173 , 971-973 (1954) doi: 10.1038/173971a0.
Hynes, TR et al. Movimiento de fragmentos de miosina in vitro:. Dominios que intervienen en la producción de fuerza Célula 48 , 953-963 (1987) Doi: 10.1016/0092-8674 (87) 90704-5.
Lehman, W., Craig, R. & Vibertt, P. movimiento de Ca2 + inducida por la tropomiosina en Limulus . filamentos delgados reveladas por la reconstrucción en tres dimensiones Naturaleza 368 , 65-67 (1994) doi: 10.1038/368065a0.
Lorand, L. «adenosina trifosfato transphosphorylase-creatina» como el factor de relajación muscular. Naturaleza 172 , 1181-1183 (1953) doi: 10.1038/1721181a0.
Spudich, JA El balanceo modelo puentes cruzados de miosina. Nature Reviews Molecular Cell Biology 2 , 387-392 (2001) doi: 10.1038/35073086.
Sliding teoría del filamento, Sarcómero, la contracción muscular, la miosina | Aprender Ciencias de Scitable.
38.969027
-0.185223













 iib.uam.es
iib.uam.es